前言
-
定义——假酶 (pseudoenzyme) 是一类具有相关家族保守序列但失去催化活性的酶。假酶不同于假基因 (pseudogenes) ,前者具有表达和功能的特征。
-
例子——假酶的重要例子包括与蛋白酶、激酶、磷酸酶、E2 泛素缀合酶和磷脂酶相关的蛋白质等。
-
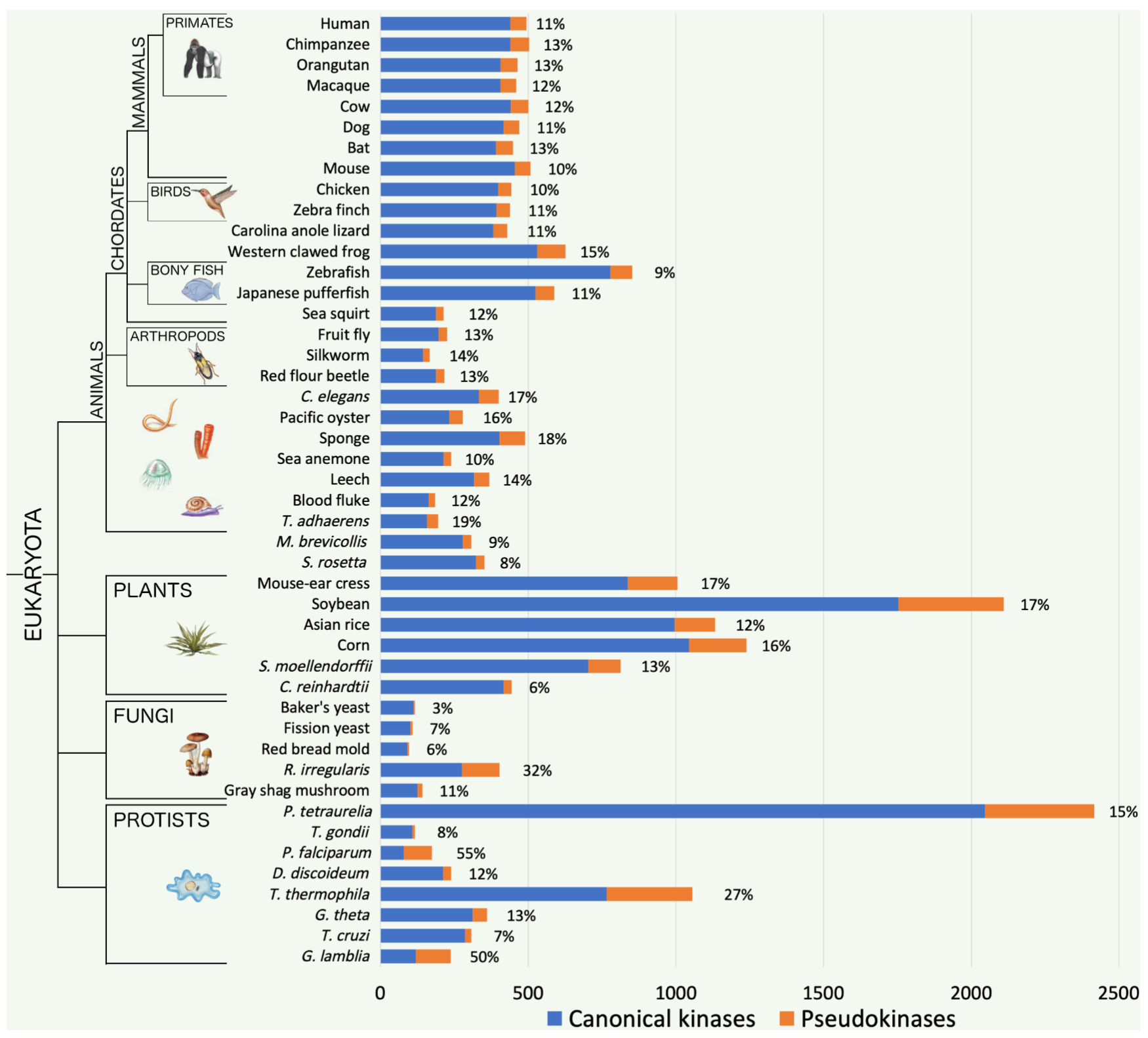
比例——假激酶
蛋白激酶存在于生命的各个领域,并在细胞信号传导中发挥不同的作用。
-
微生物:变化较大;3%~50%
-
植物:占植物激酶组的 8-17%;
-
脊椎动物:通常占激酶组含量的约 10% (假激酶组几乎总是与脊椎动物物种中激酶组的大小成正比)
-
假酶的功能
-
特点
-
假酶通常利用其与同源酶结合相同底物和辅因子的能力,并保留其物理亚细胞位置
-
假酶倾向于调节其同源酶参与的过程——可以在进化学上发挥新的功能
-
-
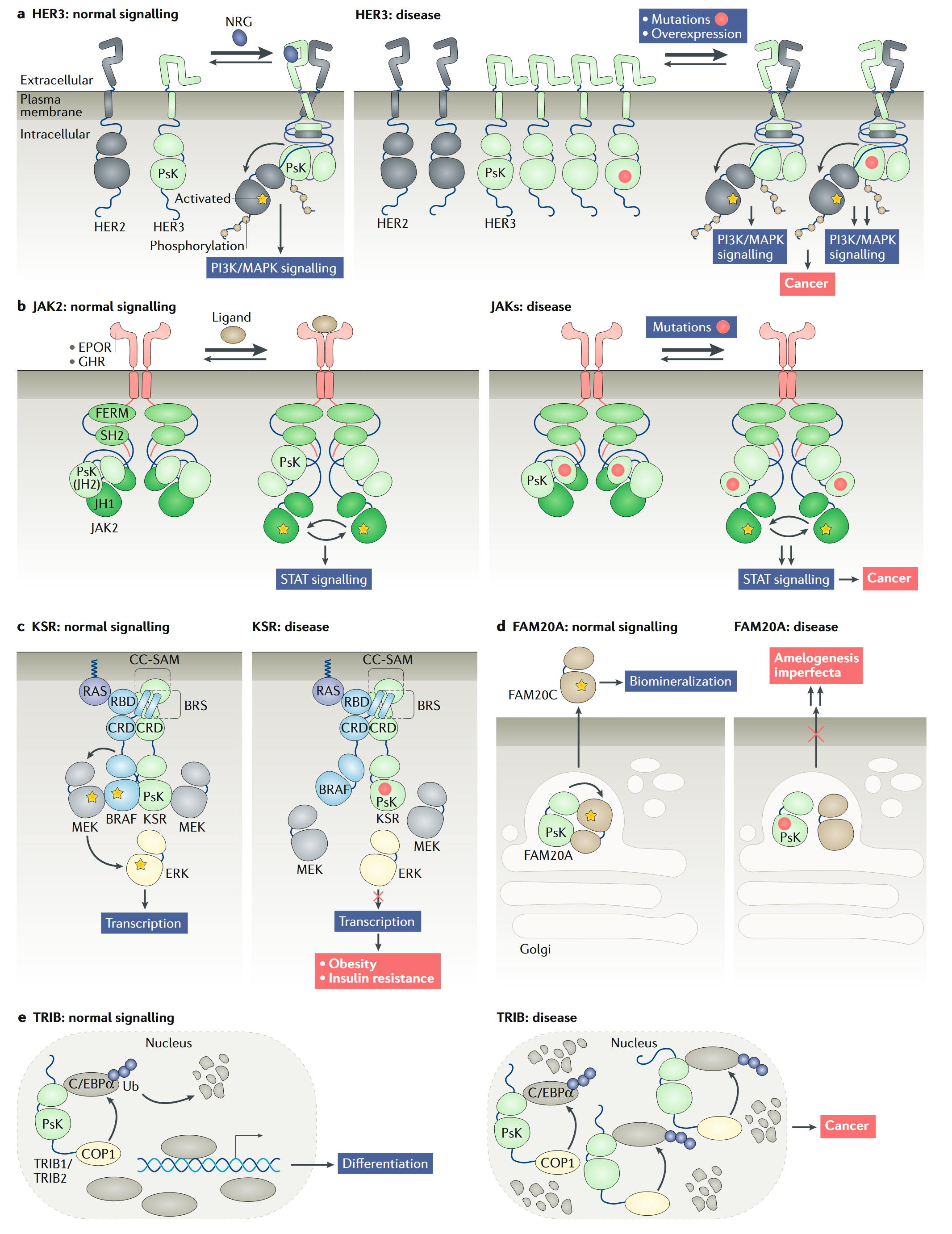
假酶发挥功能的几种形式
-
激活:与活性激酶形成异二聚体以稳定活性激酶的活性构象
- HER3——过表达、破坏其自抑制构象稳定性的突变——HER2过激活
- KSR——破坏 KSR 与 MEK 和 BRAF 的相互作用的突变——MAPK 通路激活受损
- FAM20A——突变——FAM20C 激活与分泌受损
-
抑制:变构结合活性激酶结构域,将其稳定在非活性构象
- JAK2的JH2结构域——突变——JH1结构过激活或过抑制;
-
支架:介导活性激酶的降解
- TRIB1/TRIB2——过表达—— 与C/EBPα 和 E3 泛素连接酶 COP1 相互作用增强—— C/EBPα 过度降解
-
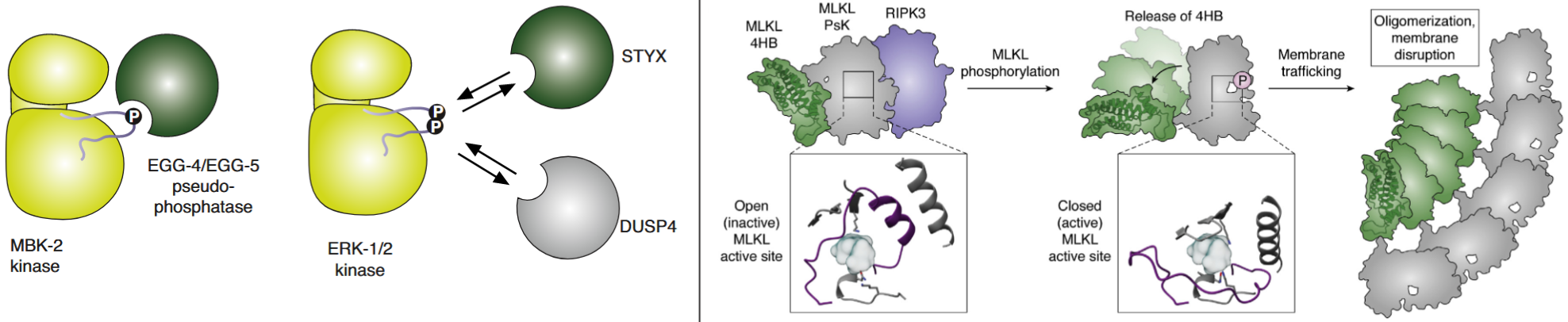
竞争底物
- 假磷酸酶 STYX 与传统磷酸酶DUSP4 竞争与 ERK1/2 激酶的结合——调控ERK1/2的活性
-
全新功能:变构形成多聚体诱发细胞凋亡
-
The mixed lineage kinase domain-like (MLKL) ——磷酸化后构象打开(分子开关)—— 形成多聚体诱发细胞凋亡
-
-
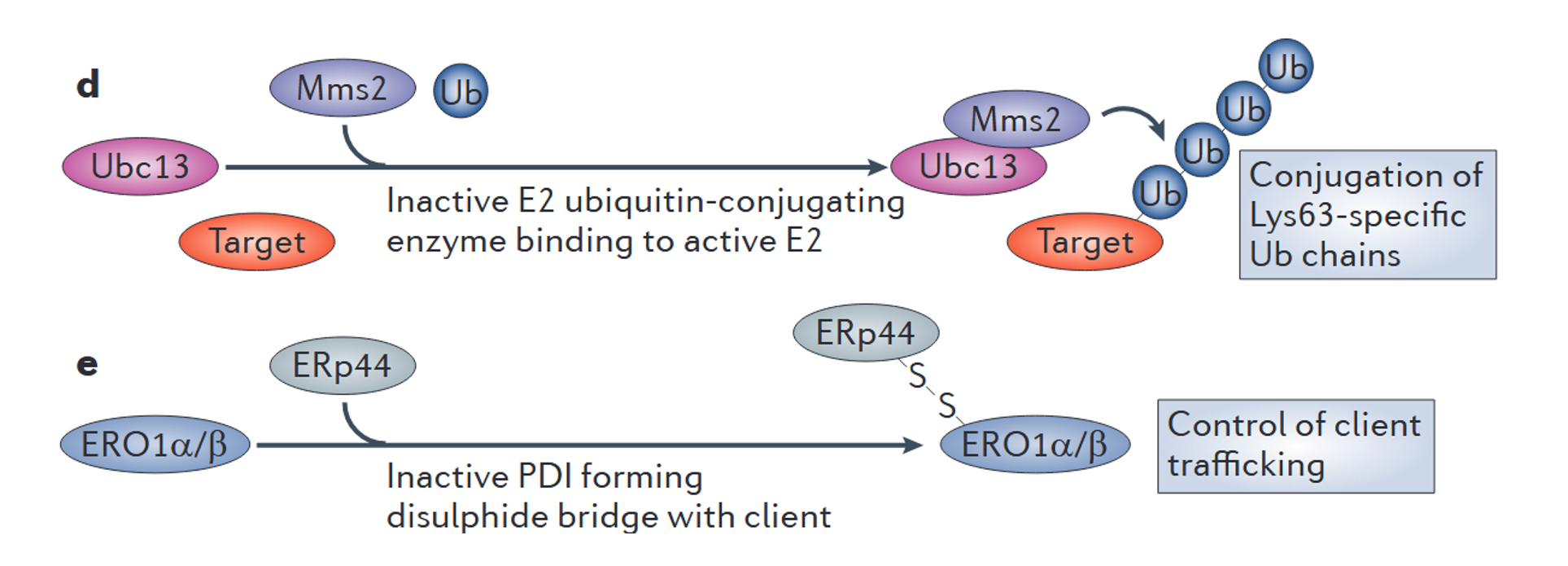
发挥祖先功能的假激酶
- Mms2(无活性的E2泛素结合酶):与另一种活性酶结合保留祖先作用的假酶
- ERp44(无活性的蛋白质二硫键异构酶 (PDI) 同源物):与祖先底物结合引发新的功能
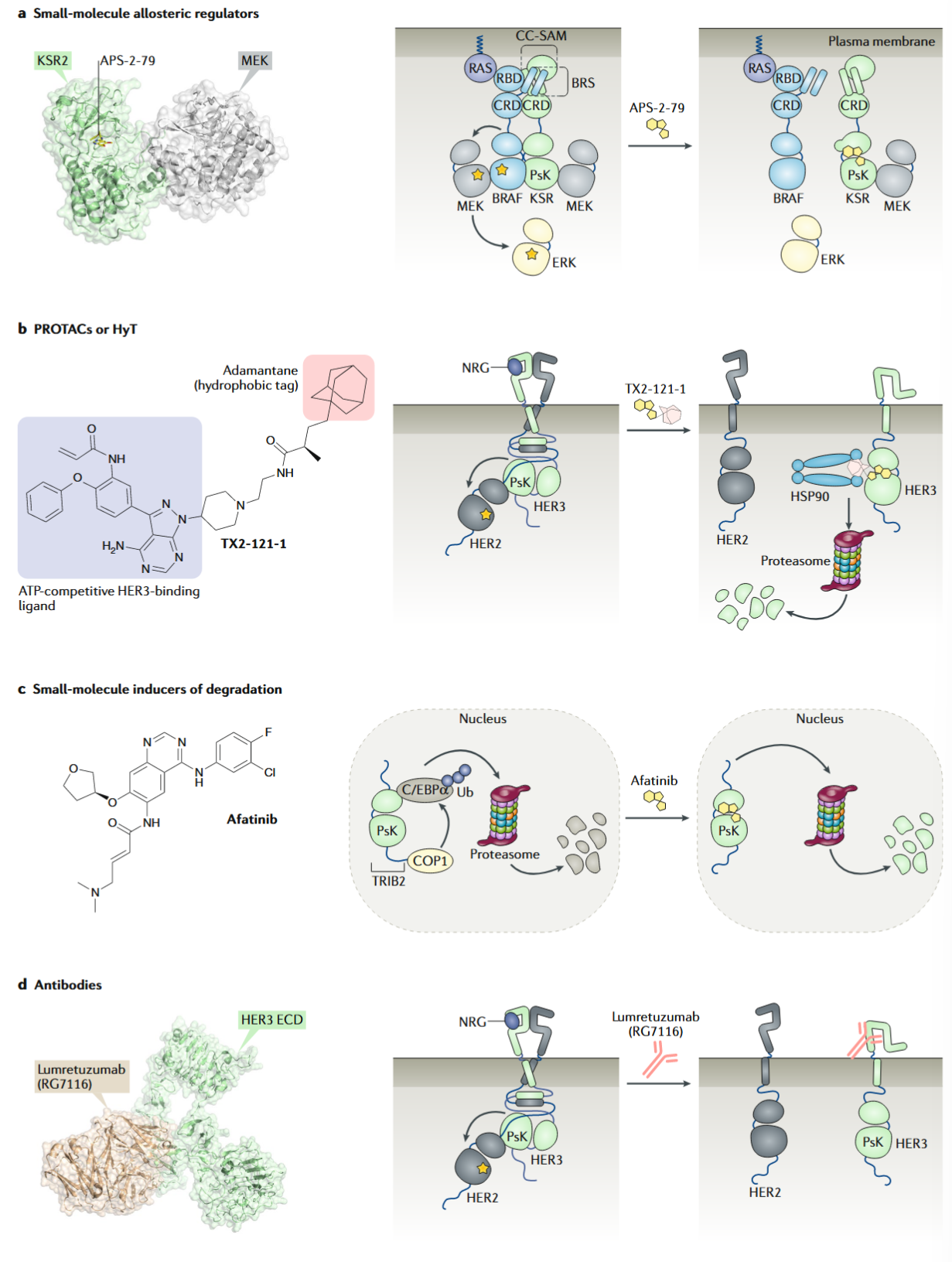
靶向假酶的药物设计
到目前为止,ATP结合位点是蛋白激酶中最“可成药”的口袋,因为它具有与小分子相互作用的进化能力。
-
抑制与活性激酶的结合——小分子、抗体:KSR2、TRIB2、HER3
-
靶向降解——PROTAC:HER3
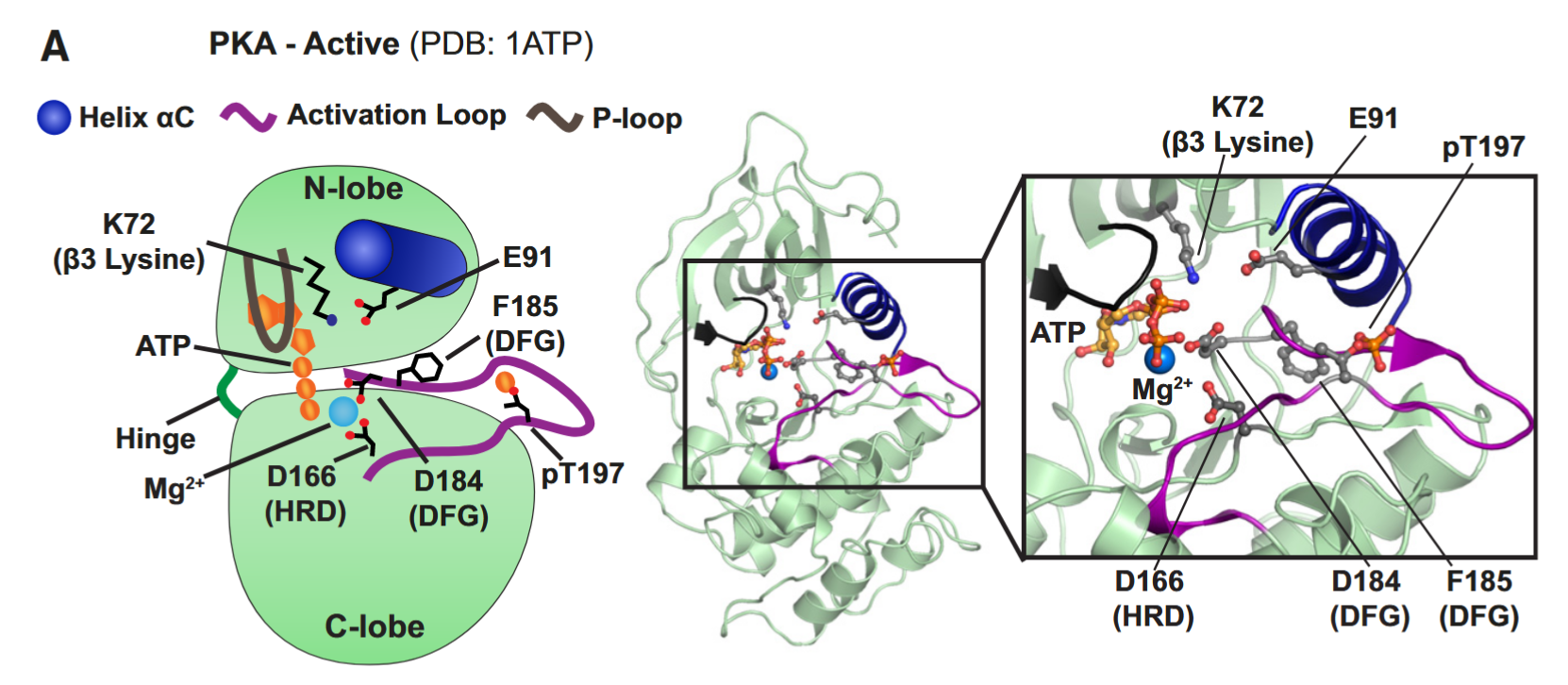
激酶的活性口袋
激酶的活性口袋由高度保守的两个叶组成,即氨基末端叶(N-叶)和C-叶,它们通过柔性铰链区连接。
与激酶在催化过程中的相对运动相关的活性位点位于 N 叶和 C 叶之间,由两个叶提供的结构元件组成。
这些运动用于调节核苷酸和底物结合、ATP水解、磷酸转移以及ADP和磷酸化底物的最终释放。
-
二价金属离子
- 特点:最典型的是 Mg2+;常与DFG位点中的天冬氨酸配位
- 功能:① 能够中和核苷酸的净负电荷,并使核苷酸能够结合到激酶活性位点;②对于 ATP 配位和磷酸转移化学也至关重要
-
保守催化残基
-
Glycine-rich-loop
-
特点:N叶中富含甘氨酸的环通常符合活性激酶中的共有序列GXGXXG。
-
功能:甘氨酸残基中不存在侧链,使得富含甘氨酸的环与 ATP 的腺苷环紧密接触,从而实现核苷酸结合和 ATP 的正确定位以进行催化。
在假激酶中,甘氨酸通常被较大的氨基酸取代,通常带负电荷,从而干扰ATP结合。
-
-
αC 螺旋:在活性构象中,螺旋αC向活性位点旋转,αC 的 Glu 与 β3 的Lys形成盐桥;
-
VAIK (Val-Ala-Ile-Lys) 基序内的赖氨酸
-
特点:位于N叶的β3链中;
-
功能:与αC 中保守的谷氨酸之间形成盐桥——协调保证ATP的正确结合
SRC/CDK-like inactive:赖氨酸与螺旋αC的盐桥在激酶的非活性构象中被破坏;螺旋αC旋转远离活性位点
-
-
DFG (Asp-Phe-Gly) 基序内的天冬氨酸
-
功能:天冬氨酸指向活性位点;协调 Mg2+离子(与 ATP 的 β-磷酸盐和 γ-磷酸盐相互作用的)
激酶的受磷酸化后,DFG会改变非活性的束缚构象并向内延伸。
-
-
HRD (His-Arg-Asp) 基序内的天冬氨酸
-
功能:参与催化过程;Asp 在 ATP 水解过程中充当催化碱基
Asp其突变将使激酶失去催化活性。
-
-
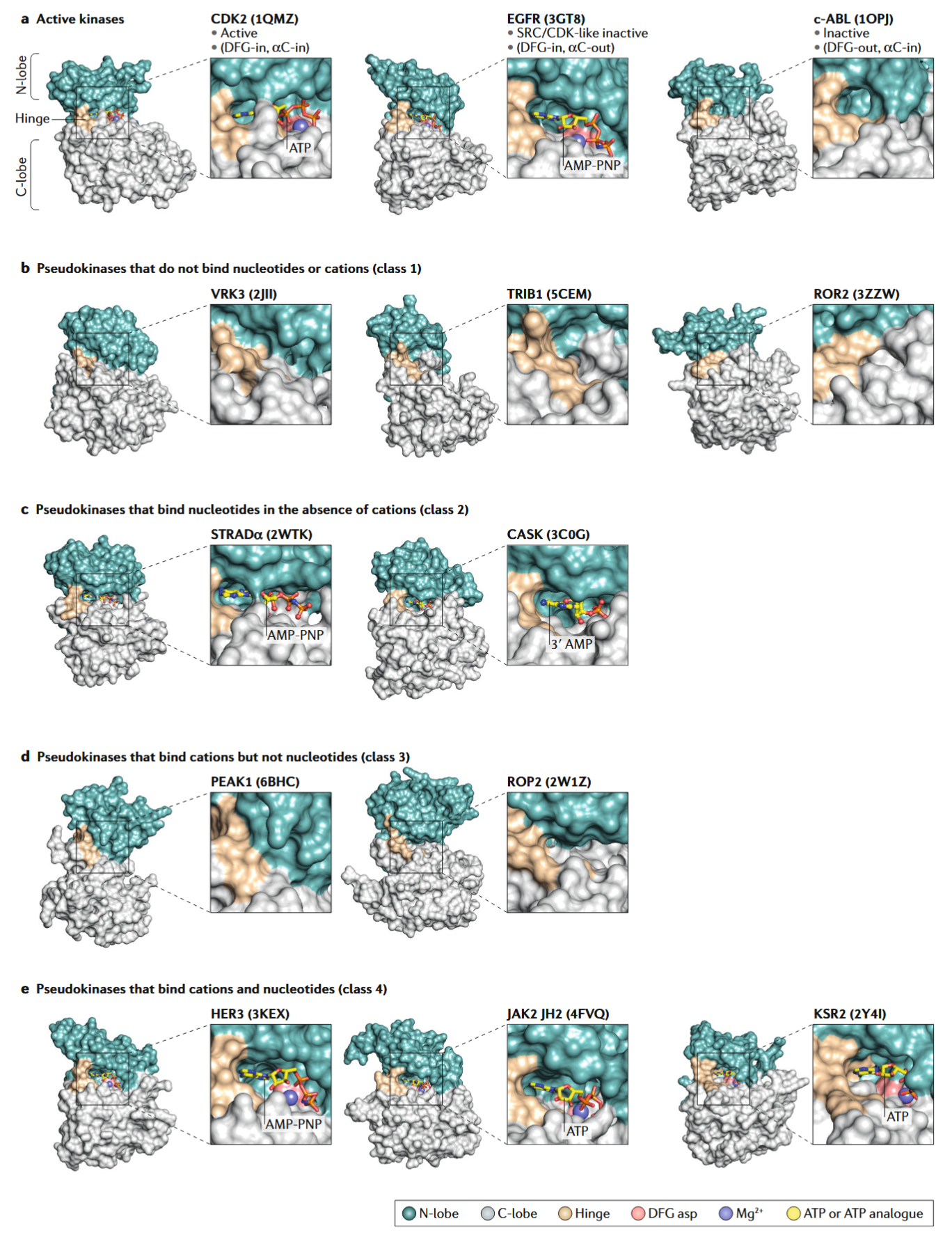
假激酶的类别
基于结构区分——ATP与金属离子的结合情况
-
Class 1 —— 不结合核苷酸或阳离子的假激酶
- 代表:TRIB1、VRK3、ROR2、Pragmin 和细菌假激酶 MviN
- 结构:核苷酸结合位点通常被疏水残基封闭——也不具有活性激酶中存在的典型可药物口袋,设计靶向药物困难
-
Class 2 —— 在没有阳离子的情况下结合核苷酸的假激酶
- 代表:STRADα、MLKL、FAM20A、CASK 和 TRIB2;
- 结构:此类中的大多数假激酶都缺乏活性激酶 DFG 基序中发现的典型金属配位天冬氨酸残基;
-
Class 3 —— 结合阳离子但不结合核苷酸的假激酶
- 代表:弓形虫蛋白棒状体蛋白 2 (ROP2) 和 PEAK1(Pragmin 的密切同源物)
- 结构
- 具有高度封闭的假活性位点,这些位点在结构上似乎与核苷酸结合不相容
- 都不具有完整的 DFG 基序;尚不清楚阳离子是否在生理环境中与 PEAK1 和 ROP2 结合
-
Class 4 —— 结合核苷酸和阳离子的假激酶
-
代表:HER3、KSR、RNase L 和 JAK 的 JH2 假激酶结构域
-
结构:具有可测量的激酶活性,通常比催化活性激酶低几个数量级。
-
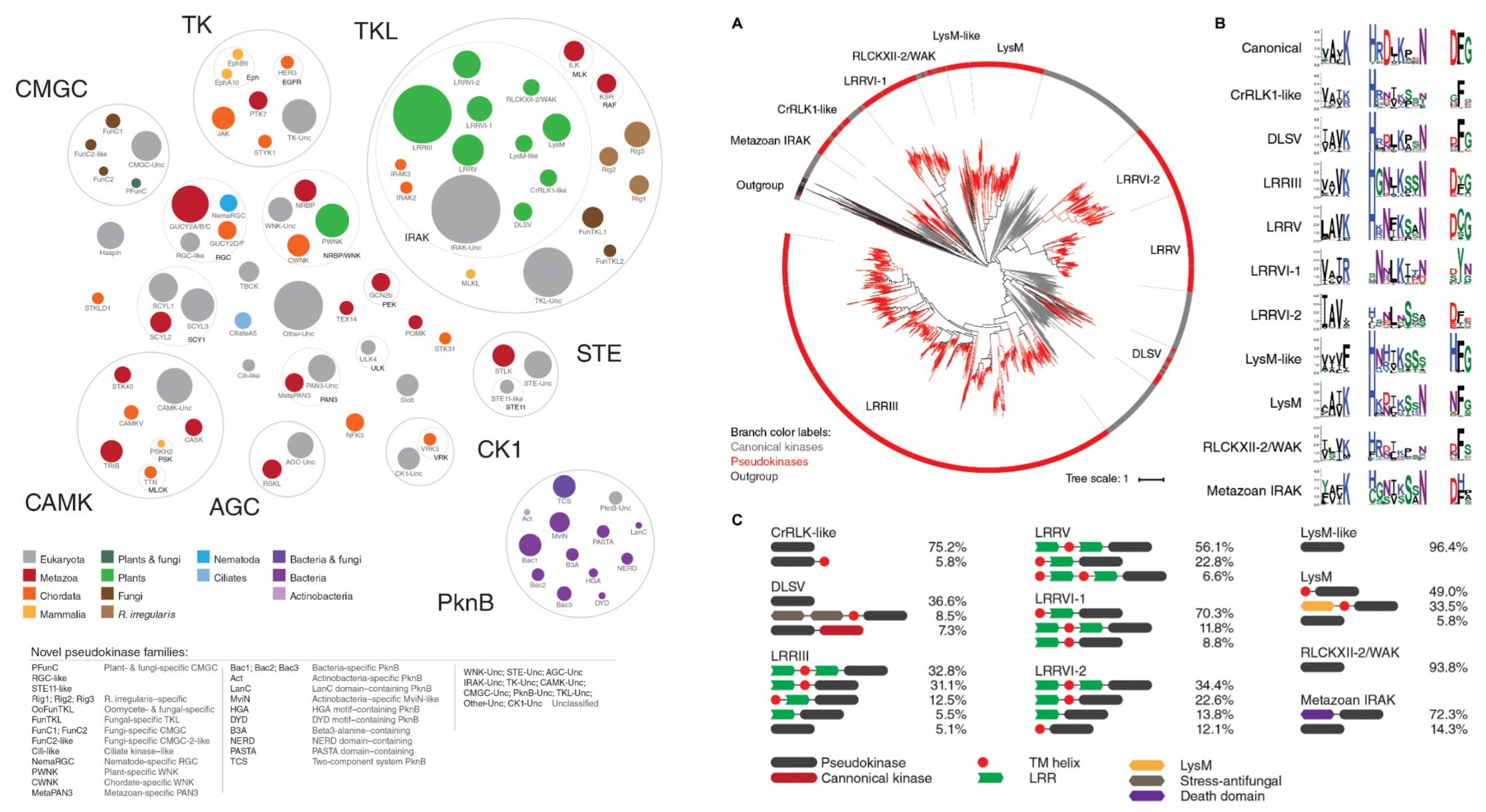
植物中的假激酶
-
酪氨酸激酶样 (TKL) 假激酶
-
植物白细胞介素-1 受体相关激酶样假激酶 (interleukin-1 receptor-associated kinase (IRAK)-like pseudokinases):TKL 组的一部分,在植物中大量扩增
-
从机制上讲,已知植物 IRAK 激酶在植物-真菌相互作用中发挥着至关重要的作用,无论是在共生相互作用期间还是在病原体防御期间,这表明真菌 TKL 激酶和假激酶的扩张可能是由于紧密的共生共同进化而产生的。
-
不同假激酶家族之间的失活激酶结构域构象通过在进化过程中被选择性限制的不同序列基序以不同的方式稳定。
-
-
一些假激酶的功能
-
LysM
-
活性LysM RLK 发挥多种传感功能:① 识别植物对真菌的防御反应中的几丁质寡糖;② 结合细菌细胞壁上的肽聚糖以帮助识别共生细菌
-
LysM 假激酶被认为通过与其他活性 LysM RLK 相互作用,有助于其适当的信号传导途径
紫花苜蓿 中的 LysM 假激酶被认为通过与活性 LysM RLK 成员相互作用来促进根瘤菌相互作用,这让人想起后生动物酪氨酸激酶和酪氨酸假激酶,例如变构调节密切相关的经典 EGFR 家族的 HER3 假激酶
-
-
DLSV:该假激酶含有与盐胁迫和抗真菌反应相关的胞外结构域
-
LRRVI-2:该假激酶与根毛形成 相关的植物钾通道 AKT2相互作用。
-
其他
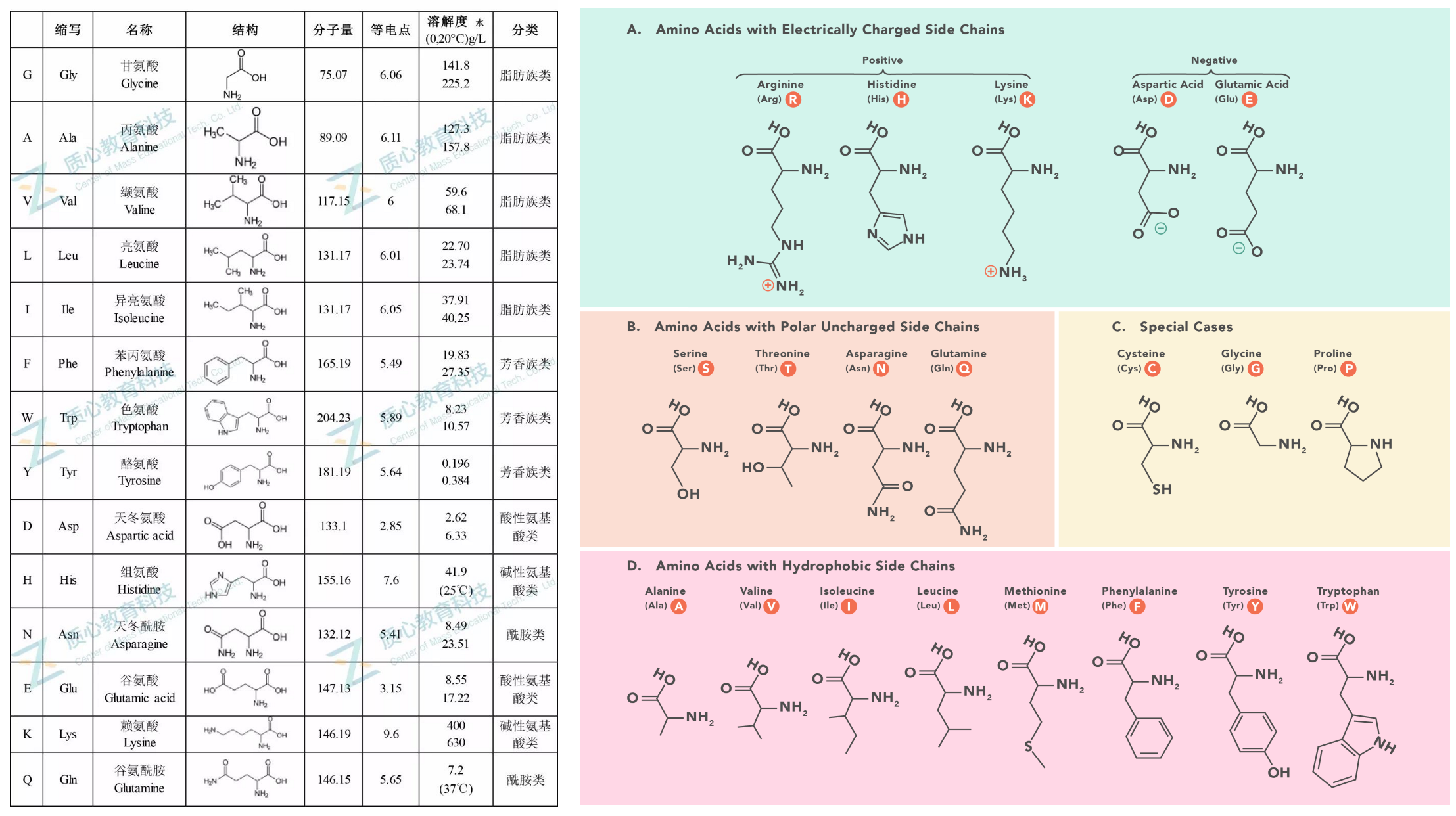
氨基酸表
参考文献
- Adrain C, Freeman M. New lives for old: evolution of pseudoenzyme function illustrated by iRhoms. Nat Rev Mol Cell Biol, 2012, 13(8): 489-498.
- Kung JE, Jura N. Structural basis for the non-catalytic functions of protein kinases. Structure, 2016, 24(1): 7-24.
- Murphy JM, Mace PD, Eyers PA. Live and let die: insights into pseudoenzyme mechanisms from structure. Curr Opin Struct Biol, 2017, 47: 95-104.
- Kung JE, Jura N. Prospects for pharmacological targeting of pseudokinases. Nat Rev Drug Discov, 2019, 18(7): 501-526.
- Kwon A, Scott S, Taujale R, Yeung W, Kochut KJ, Eyers PA, Kannan N. Tracing the Origin and Evolution of Pseudokinases Across the Tree of Life. Sci Signal, 2019, 12(578): eaav3810.
- Ribeiro AJM, Das S, Dawson N, Zaru R, Orchard S, Thornton JM, Orengo C, Zeqiraj E, Murphy JM, Eyers PA. Emerging concepts in pseudoenzyme classification, evolution, and signaling. Sci Signal, 2019, 12(594): eaat9797.
- Mace PD, Murphy JM. There’s more to death than life: Noncatalytic functions in kinase and pseudokinase signaling. J Biol Chem, 2021, 296: 100705.